Pythonで予測モデルを作るときの大まかな流れの雛形みたいなやつ(自己流なので正しいかはわかりませんが…)をメモしていきます。
1. データの読み込みと確認
Boston Housingデータを使います。サンプルサイズが500程度なのでかなり小さいですが,まぁ例なのでご了承ください。
import pandas as pd # データの読み込みfrom sklearn.datasets import load_boston boston = load_boston() boston_df = pd.concat([pd.DataFrame(boston['data'], columns = boston['feature_names']), pd.Series(boston['target']).rename('MEDV')],axis=1) boston_df.head() # print(boston.DESCR) # データの詳細
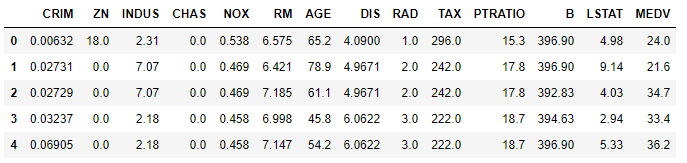
データの確認
変数の意味は以下のような感じらしいです。
- CRIM:一人あたり犯罪率
- ZN:25,000平方フィート以上の住宅区画の割合
- 25,000平方フィート=2322.576平方メートル
- INDUS:町ごとの非小売業の土地面積の割合
- CHAS:チャールズ川ダミー(川沿いなら1,そうでないなら0)
- NOX:一酸化窒素濃度(1000万分率)
- RM:1戸あたりの平均部屋数
- AGE:1940年よりも前に建てられた持家住宅の割合
- DIS:ボストンの主な5つの雇用圏までの重み付き距離
- RAD:幹線道路へのアクセス可能性の指標
- TAX:1万ドルあたりの固定資産税の最大税率
- PTRATIO:町ごとの生徒-教師の比率
- B:[tex: 1000(Bk-0.63)2] , ここで
は町ごとの黒人の割合
- LSTAT:低所得者の割合
- MEDV:持ち家住宅の価格の中央値(単位:1000ドル)
一般的に予測の目的変数はMEDVらしいので,今回もそうします。
データ間の関係を確認
データの可視化(単変量)
pandas_profilingでヒストグラムなどを描いてざっくり可視化してもらいます。
# pandas_profilingによるお手軽なデータ可視化import pandas_profiling as pdp report = pdp.ProfileReport(boston_df) report.to_file(outputfile="report.html")
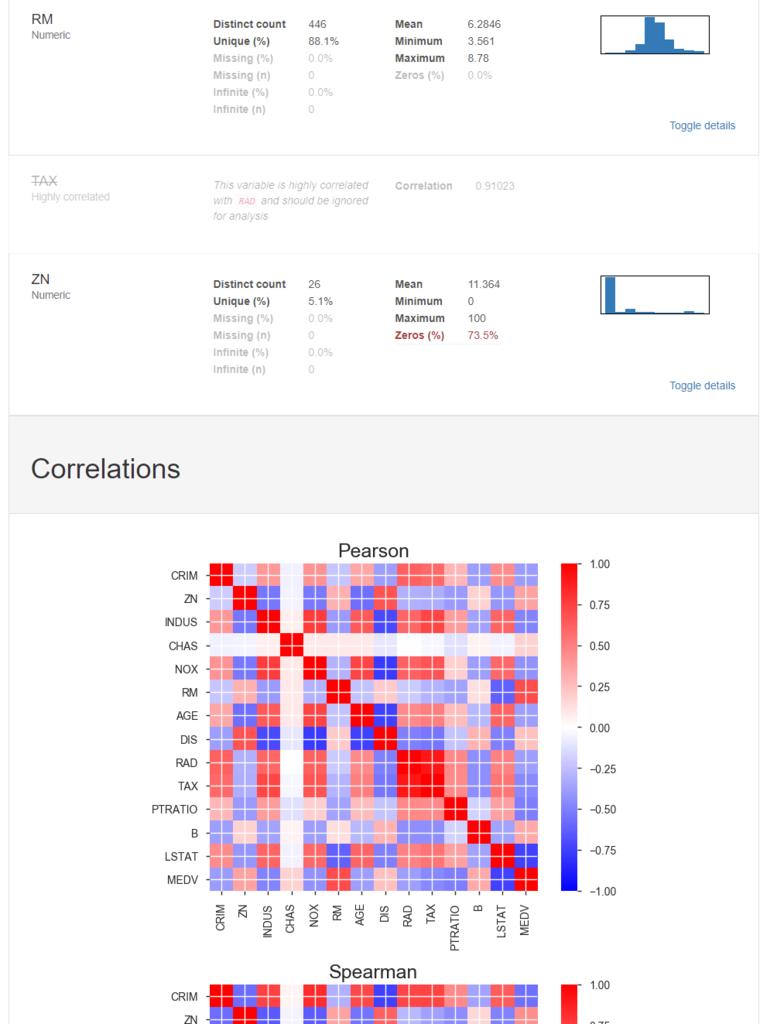
データの可視化(多変量)
相関行列・ヒートマップ等
2変量の間の関係の強さを見ていきたいと思います。
ヒートマップはpandas_profilingが出してくれていたので,数字で見てみます。
# 相関係数の絶対値でソート boston_df.corr()[['MEDV']].abs().sort_values('MEDV')
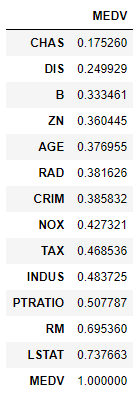
線形な相関関係の強さでいうとLSTAT(低所得者の割合)やRM(1戸あたりの平均部屋数)が高いみたいですね。
グラフ
相関係数(2変数間の直線的な関係を示す)が高くても,実際の分布が直線的(線形)かそうでない(非線形)かはプロットしてみないとわかりません。
実際にプロットしてみると非線形な関係でした
import matplotlib.pyplot as plt % matplotlib inline # Jupyter Notebookで描くときのみ plt.scatter(x = boston_df['LSTAT'], y = boston_df['MEDV']) plt.xlabel('LSTAT') plt.ylabel('MEDV')
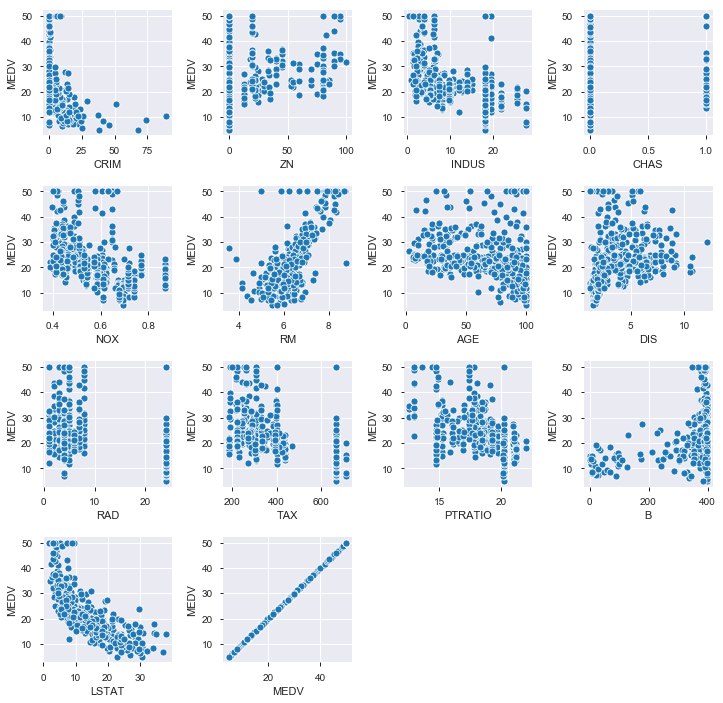
他の変数とMEDVの関係も散布図で見てみます。やはり非線形な関係にある変数が多いです。
# MEDVとその他全変数との散布図 plt.figure(figsize=(12,12)) plt.subplots_adjust(wspace=0.4, hspace=0.4) # subplotの余白 i = 1for var in boston_df.columns: plt.subplot(4, 4, i) # 4×4のグリッドに順番にplotしていく sns.scatterplot(x = var, y = 'MEDV', data = boston_df) i = i + 1 plt.show()
2. 前処理
※実際の分析ではここからが重要なパートなのですが,今回のデータだとあんまりいじり甲斐がないのでこのあたりは省略します。
ドメイン知識の収集・整理
データを正しく扱うために,分析対象に関する知識を仕入れていきます。
特徴量エンジニアリング
データを集計した結果およびドメイン知識から,既存のデータから新たな特徴量を生み出したり,外部データを持ってきたりします。
データのクリーニング
欠損値がある場合,補完を行います。(今回のデータは欠損値なし)
# 欠損値の確認 boston_df.isnull().sum()
CRIM 0 ZN 0 INDUS 0 CHAS 0 NOX 0 RM 0 AGE 0 DIS 0 RAD 0 TAX 0 PTRATIO 0 B 0 LSTAT 0 MEDV 0 dtype: int64
もし欠損があった場合,以下の手法を使うのが手っ取り早いです
- fillna(穴埋め):ある変数のすべての欠損値に,何らかの一つの値(その変数の中央値や最頻値など)を代入する
fillna()はDataFrameに対しても,Seriesに対しても使えます。- https://pandas.pydata.org/pandas-docs/stable/generated/pandas.DataFrame.fillna.html
- interpolate(補間):欠損のある変数に対する予測モデルを作り,算出した予測値で代入する方法
データの分割
# データの分割 X = boston_df.drop(['MEDV'],axis=1) y = boston_df['MEDV'] # trainデータ, testデータへの分割from sklearn.model_selection import train_test_split X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=0)
3. 予測
3つの予測モデルを使ってアンサンブルしてみます。
ElasticNetによる予測
パラメータチューニング
# GridSearchによるパラメータチューニングfrom sklearn.linear_model import ElasticNet from sklearn.model_selection import GridSearchCV from sklearn.metrics import mean_squared_error # grid search parameter_range = [{'alpha': [0.00001, 0.0001, 0.001, 0.01, 0.1, 1], 'l1_ratio' : [0.001, 0.01, 0.1, 1]}] model = GridSearchCV(estimator = ElasticNet(normalize=True, max_iter=10000), param_grid = parameter_range, cv = 10, scoring = 'neg_mean_squared_error') model.fit(X_train, y_train) # best estimator best_model = model.best_estimator_
予測精度の評価
Cross Validationによる予測精度の評価
- 誤差関数の参考:3.3. Model evaluation: quantifying the quality of predictions — scikit-learn 0.20.2 documentation
# Cross Validationによる予測精度の評価from sklearn.model_selection import cross_validate from sklearn.metrics import mean_squared_error, r2_score, make_scorer # 誤差関数の定義defroot_mean_squared_error(y_true, y_pred): return np.sqrt(mean_squared_error(y_true, y_pred)) defmedian_absolute_error_rate(y_true, y_pred): return np.median(np.absolute(y_true - y_pred) / y_true) # Cross Validationで使う誤差関数のセット scoring = {"r2": "r2", "RMSE":make_scorer(root_mean_squared_error), "MER":make_scorer(median_absolute_error_rate)} # cross_validate scores = cross_validate(best_model, X_train, y_train, cv = 10, scoring = scoring, return_train_score=False) for key,value in scores.items(): print("{}:{:.3g}+/-{:.3g}".format(key, value.mean(), value.std())) # モデルを格納 best_model_EN = best_model # 結果を格納 cv_accuracy_EN = pd.DataFrame(scores)[['test_r2','test_RMSE','test_MER']].mean()
test_r2:0.732+/-0.106 test_RMSE:4.56+/-0.758 test_MER:0.115+/-0.0163
testデータに対する予測精度の確認
学習させずにとっておいたtestデータに対する予測精度を評価します。
# 予測値の算出 y_pred = pd.Series(best_model.predict(X_test)) y_pred.name = 'predicted'# 誤差を算出 y_true = y_test.reset_index(drop=True) print('R2:', '{:.3g}'.format(r2_score(y_true, y_pred))) print('RMSE:', '{:.3g}'.format(root_mean_squared_error(y_true, y_pred))) print('MER:', '{:.3g}'.format(median_absolute_error_rate(y_true, y_pred)))
R2: 0.586 RMSE: 5.81 MER: 0.133
regplot
# 予測-実測プロット
sns.regplot(y_true, y_pred)

RandomForestによる予測
パラメータチューニング
# GridSearchによるパラメータチューニングfrom sklearn.ensemble import RandomForestRegressor from sklearn.model_selection import GridSearchCV from sklearn.metrics import mean_squared_error # grid search parameter_range = [{'n_estimators': [20, 50, 100, 200], 'max_depth' : [5, 10, 50, 100, 200]}] model = GridSearchCV(estimator = RandomForestRegressor(), param_grid = parameter_range, cv = 10, scoring = 'neg_mean_squared_error') model.fit(X_train, y_train) # best estimator best_model = model.best_estimator_
Cross Validationによる予測精度の評価
ここのコードは上のと同じなので結果だけ。
test_r2:0.863+/-0.0899 test_RMSE:3.17+/-0.855 test_MER:0.069+/-0.0178
testデータに対する予測精度の確認
R2: 0.752 RMSE: 4.49 MER: 0.0779
特徴量重要度の確認
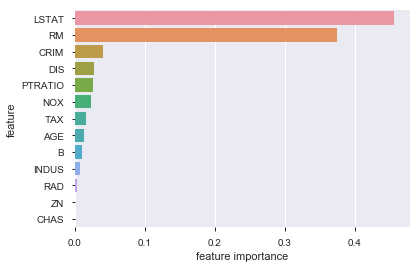
# feature importances fi = best_model.feature_importances_ fi_df = pd.DataFrame({'feature': list(X_train.columns), 'feature importance': fi[:]}).sort_values('feature importance', ascending = False) fi_df
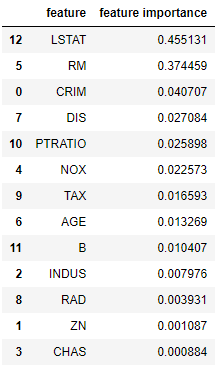
sns.barplot(fi_df['feature importance'],fi_df['feature'])
合計が100%になるように累積分布にして図を描いたらわかりやすいかも,と思ったので描いてみます。
# 累積 feature importance sns.lineplot(x = fi_df['feature'], y = fi_df['feature importance'].cumsum(), marker="o", sort=False) plt.ylim(0) plt.hlines(y = 1, xmin = 0, xmax = 12) # y = 1 の水平線 plt.xticks(rotation=90)
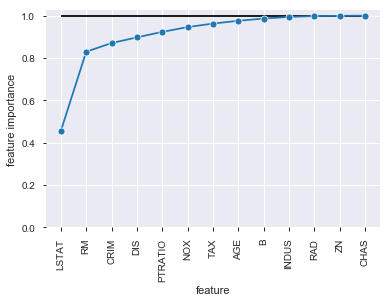
ZNあたりからはほとんど予測に寄与していなさそうにも思えます。
いくつかの変数を除外したモデルを試しに作ってみます。
Feature Importanceに基づく特徴量選択
Feature Importanceの相対度数が0.001以上の特徴量だけを使って再度学習させてみます。
「Feature ImportanceがXX以上の特徴量だけを使う」という処理は
use_features = fi_df.loc[fi_df['feature importance'] >= XX,'feature'] X_train2 = X_train[use_features]
と書くこともできますし,
from sklearn.feature_selection import SelectFromModel selector = SelectFromModel(best_model, threshold = XX, prefit = True) X_selected = selector.transform(X_train)
と書くこともできます。
arrayで返ってくるSelectFromModelよりは自分でdf.loc[]で選んだほうが,選択した特徴量の把握がやりやすい気がします。
今回はFeature Importanceの”相対度数”を使うのでSelectFromModelは使えないためdf.loc[]で選びます。
# feature importanceの相対度数が0.01以上の特徴量を使う use_features_RF = fi_df.loc[fi_df['FI_ratio'] >= 0.001,'feature'] X_train2 = X_train[use_features_RF]
特徴量を減らした以外は同様に予測させた結果がこちら。
予測精度の評価
Cross Validationによる予測精度の評価
test_r2:0.863+/-0.0881 test_RMSE:3.17+/-0.774 test_MER:0.0756+/-0.018
testデータに対する予測精度の確認
R2: 0.726 RMSE: 4.72 MER: 0.0822
特徴量を絞ったことで予測精度が少し悪化しました。
後のアンサンブル用に,特徴量を絞らなかった方のモデルとその予測精度の評価結果を格納しておきます。
best_model_RF = best_model cv_accuracy_RF = pd.DataFrame(scores)[['test_r2','test_RMSE','test_MER']].mean()
LGBMによる予測
パラメータチューニング
import lightgbm as lgb # grid search parameter_range = {'num_leaves': [5, 10, 20], 'learning_rate': [0.01, 0.1, 1], 'n_estimators': [100, 200, 500], 'max_depth': [2, 4, 6, 8, 10, -1]} model = GridSearchCV(estimator = lgb.LGBMRegressor(), param_grid = parameter_range, cv = 10, scoring = 'neg_mean_squared_error') model.fit(X_train, y_train) # best estimator best_model = model.best_estimator_
予測精度の評価
Cross Validationによる予測精度の評価
test_r2:0.889+/-0.0689 test_RMSE:2.83+/-0.5 test_MER:0.0755+/-0.0138
testデータに対する予測精度の確認
R2: 0.714 RMSE: 4.82 MER: 0.0965
特徴量重要度の確認
RandomForestと同様に.feature_importances_で特徴量重要度を見ることができます。
# feature importances fi = best_model.feature_importances_ fi_df = pd.DataFrame({'feature': list(X_train.columns), 'feature importance': fi[:]}).sort_values('feature importance', ascending = False) fi_df


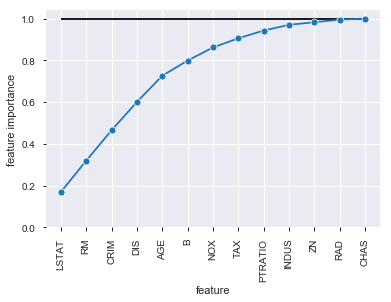
特徴量選択
feature importanceの相対度数が0.01に満たない特徴量(CHAS)を落として再度予測をさせてみます。
Cross Validationによる予測精度の評価
test_r2:0.89+/-0.0743 test_RMSE:2.81+/-0.485 test_MER:0.0653+/-0.00994
testデータに対する予測精度の確認
R2: 0.759 RMSE: 4.43 MER: 0.0893
LGBMは特徴量を絞ったほうが予測精度が良くなりました。
後のアンサンブル用にモデルと精度評価結果を格納しておきます。
best_model_LGBM = best_model cv_accuracy_LGBM = pd.DataFrame(scores)[['test_r2','test_RMSE','test_MER']].mean()
4. 予測モデルのアンサンブル
3つの予測モデルから予測値を得ることができました。
# 予測値の算出
y_pred_EN = pd.Series(best_model_EN.predict(X_test))
y_pred_RF = pd.Series(best_model_RF.predict(X_test))
y_pred_LGBM = pd.Series(best_model_LGBM.predict(X_test[use_features_LGBM]))
これらの予測値を統合し,より良い予測値にしたいと思います。
もし予測モデルたちがどれも同程度に予測性能が良いのであれば単純に平均(一様な重み付け)をとってもいいのですが,今回は予測精度に差があるため,適切な重み付けを推定することを考えていきます。
今回は2つのアプローチを試してみます。 1. 誤差の指標に応じた重み付け 2. OLSで重みを推定する
誤差の指標に応じた重み付け
cross_validateで評価した各モデルの誤差の指標を加工して重みの値にします。
# cross validationの結果 cv_accuracy_df = pd.concat([cv_accuracy_EN, cv_accuracy_RF, cv_accuracy_LGBM],axis=1) cv_accuracy_df.columns = ['EN','RF','LGBM'] cv_accuracy_df
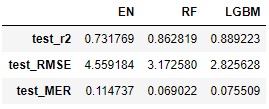
決定係数を基準にする場合
決定係数のように「指標の値が高いほど予測精度が高いことを示す」ような指標なら,
重みの合計が1になるように決定係数の合計で除して,それを重みに使います。
# r2を基準にする場合 accuracy_scores = cv_accuracy_df.loc['test_r2',] ## 合計が1になるように変換 accuracy_scores = accuracy_scores / accuracy_scores.sum() accuracy_scores
EN 0.294615 RF 0.347377 LGBM 0.358007 Name: test_r2, dtype: float64
これが重みになります。
これで予測値を統合した場合のtestデータとの誤差は
# アンサンブルした予測値の算出 y_pred_ens = accuracy_scores[0] * y_pred_EN + accuracy_scores[1] * y_pred_RF + accuracy_scores[2] * y_pred_LGBM # 誤差を算出 y_true = y_test.reset_index(drop=True) print('R2:', '{:.3g}'.format(r2_score(y_true, y_pred_ens))) print('RMSE:', '{:.3g}'.format(root_mean_squared_error(y_true, y_pred_ens))) print('MER:', '{:.3g}'.format(median_absolute_error_rate(y_true, y_pred_ens)))
R2: 0.741 RMSE: 4.6 MER: 0.0916
こうなります。
RMSEを基準にする場合
RMSEやMERなど「指標の値が低いほど予測精度が高いことを示す」ような指標は逆数にしてから同様の処理を行います。
# RMSEを基準にする場合## 逆数を使う accuracy_scores = 1 / cv_accuracy_df.loc['test_RMSE',] ## 合計が1になるように変換 accuracy_scores = accuracy_scores / accuracy_scores.sum() accuracy_scores
EN 0.246879 RF 0.354779 LGBM 0.398342 Name: test_RMSE, dtype: float64
予測の誤差はこうなりました。
R2: 0.746 RMSE: 4.54 MER: 0.0888
また,MERを基準にしたものも同様に試しましたが,予測の誤差は
R2: 0.747 RMSE: 4.54 MER: 0.0906
というもので,このアプローチではいずれもLGBM単体の予測精度に劣る結果になりました。
OLSで重みを推定
各予測モデルでtrainデータでの予測値を算出し,それらの線形和でtrainデータの実測値を近似するOLSを回してみます。
# アンサンブルで予測値の重み付け和を作る## 各予測モデルのtrainデータの予測値の算出 y_train_pred_EN = pd.Series(best_model_EN.predict(X_train)) y_train_pred_RF = pd.Series(best_model_RF.predict(X_train)) y_train_pred_LGBM = pd.Series(best_model_LGBM.predict(X_train[use_features_LGBM])) pred_df = pd.concat([y_train_pred_EN, y_train_pred_RF, y_train_pred_LGBM],axis=1) ## 最適な重みをOLSで推定するfrom sklearn.linear_model import LinearRegression lm = LinearRegression(fit_intercept=False) lm.fit(pred_df, y_train) lm.coef_
array([-0.07140172, 0.23783679, 0.83375187])
この係数も合計が1になるように調整して使います
# 係数の合計が1になるように調整 coef = lm.coef_ / lm.coef_.sum() # アンサンブルした予測値の算出 y_pred_ens = coef[0] * y_pred_EN + coef[1] * y_pred_RF + coef[2] * y_pred_LGBM
R2: 0.766 RMSE: 4.37 MER: 0.0856
だいぶ予測精度が良くなりました。
LGBMの予測精度が
R2: 0.759 RMSE: 4.43 MER: 0.0893
だったので,それよりも良い予測精度になりました。
今回はデータセットのサンプル数が少なかったので,もっとしっかりした規模のデータを扱うときもOLSを使ったほうが良い結果になるのかはあとで試してみたいですね…